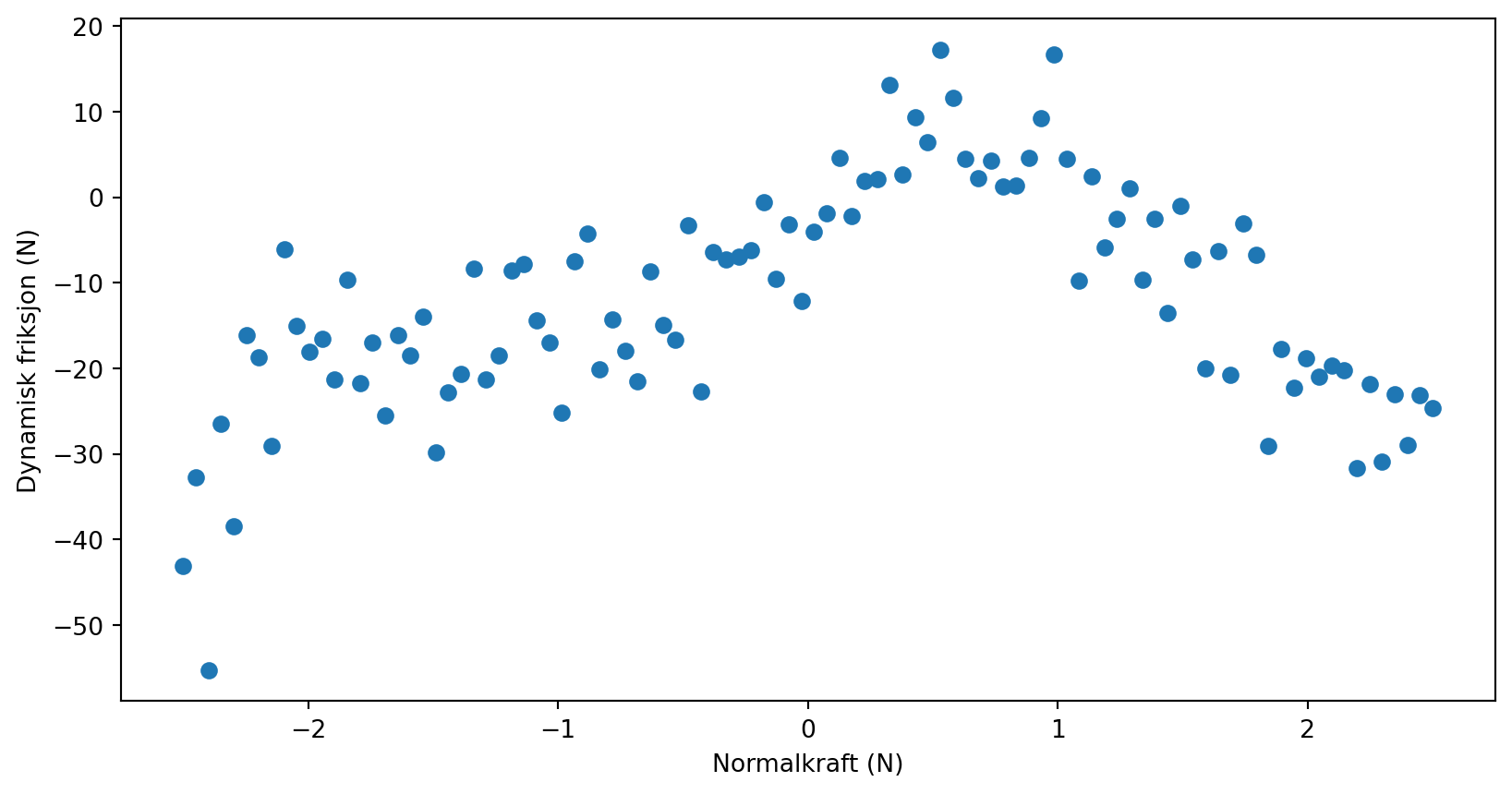
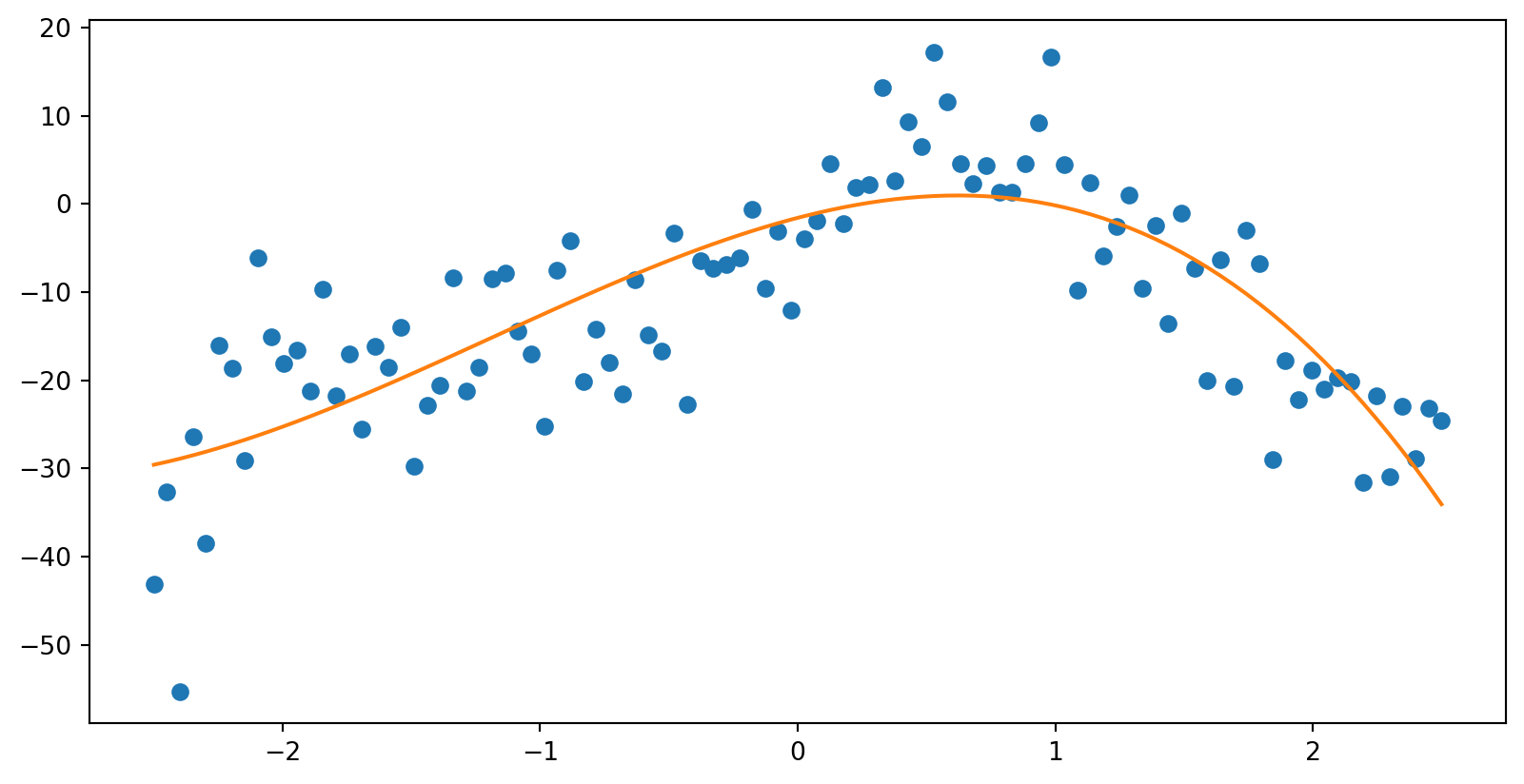
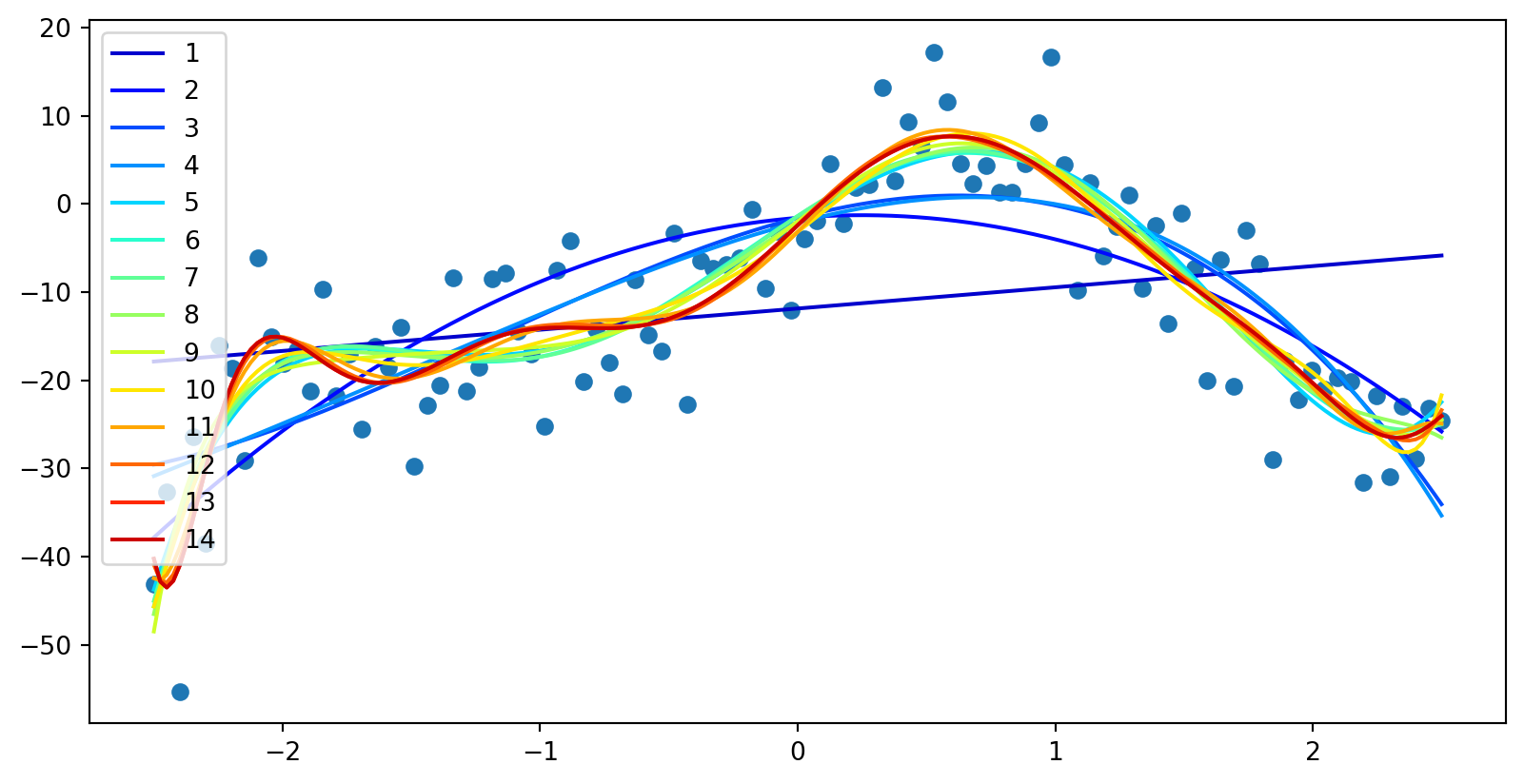
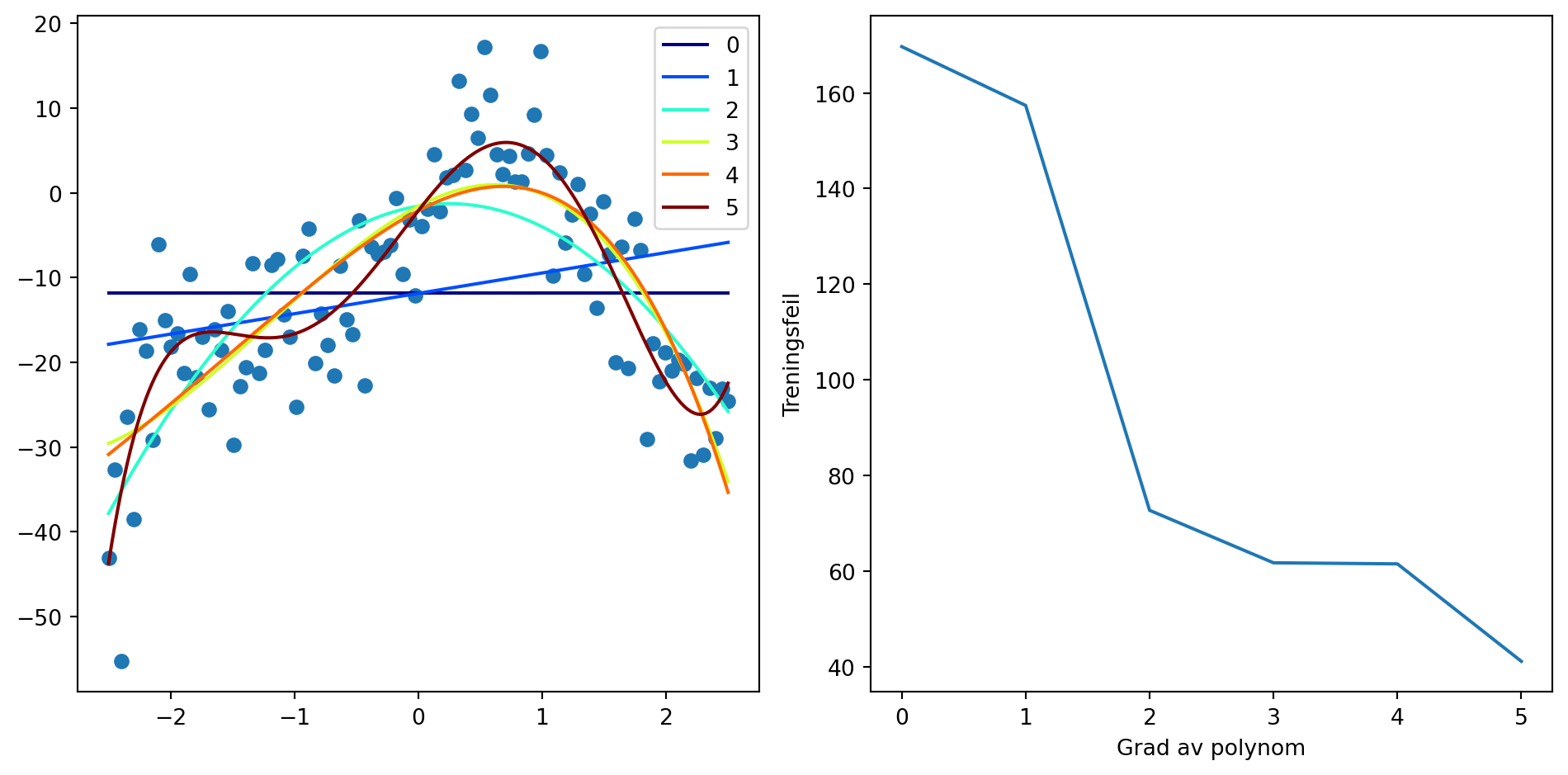
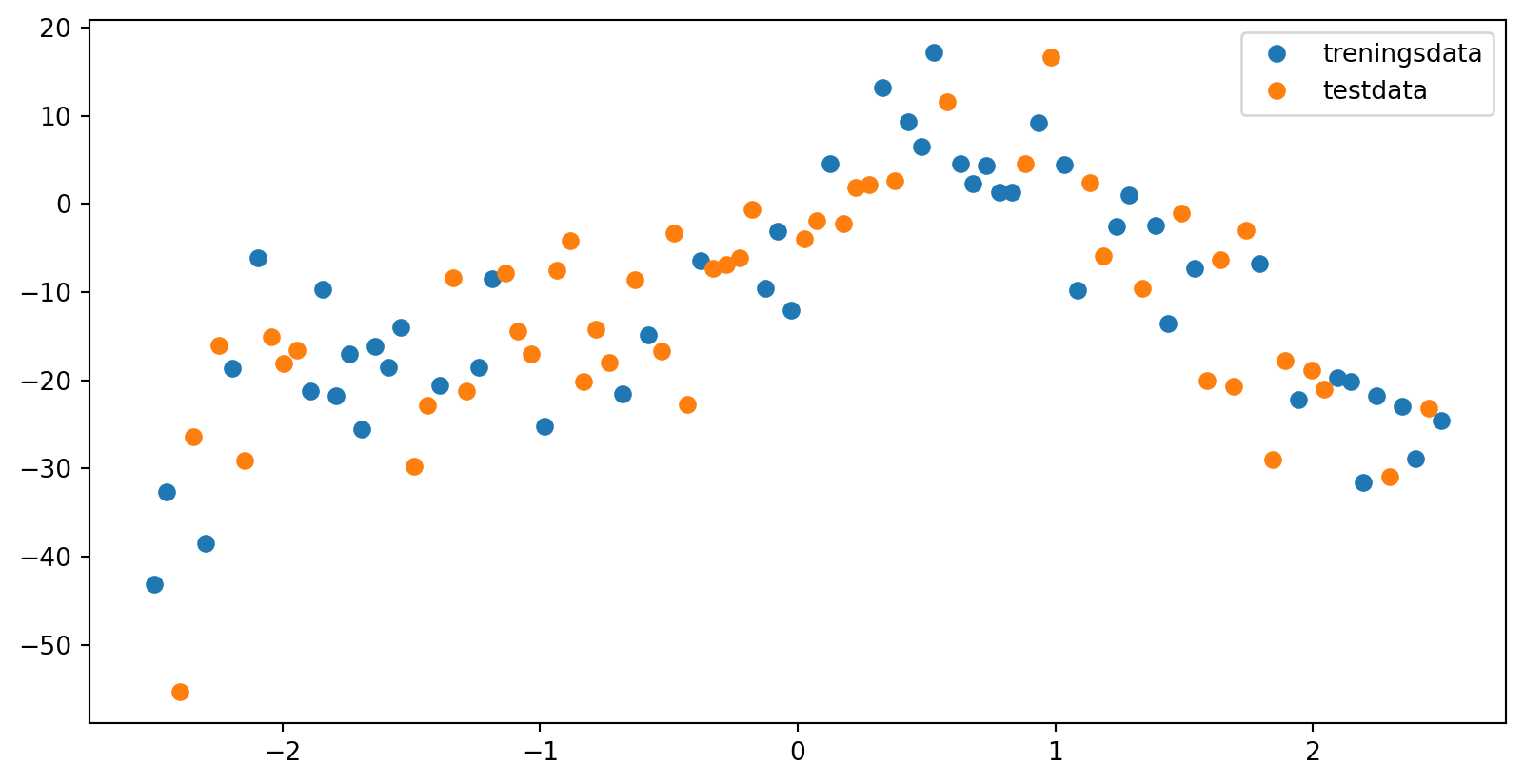
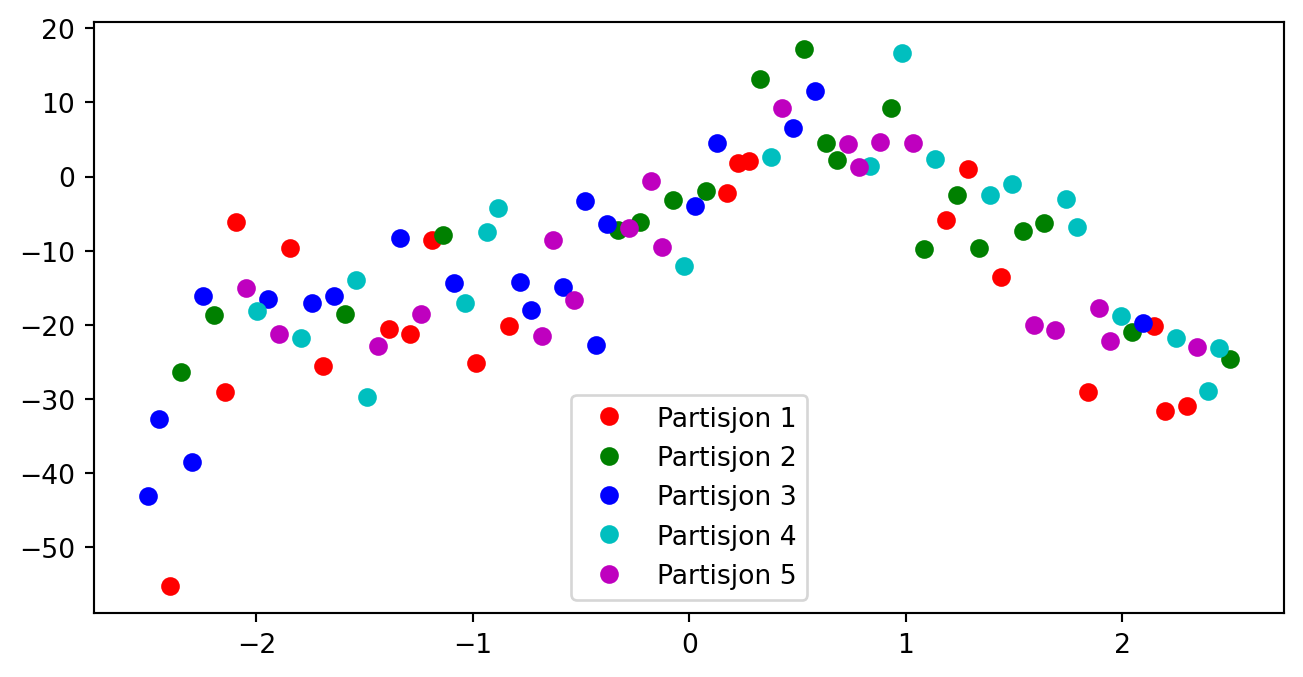
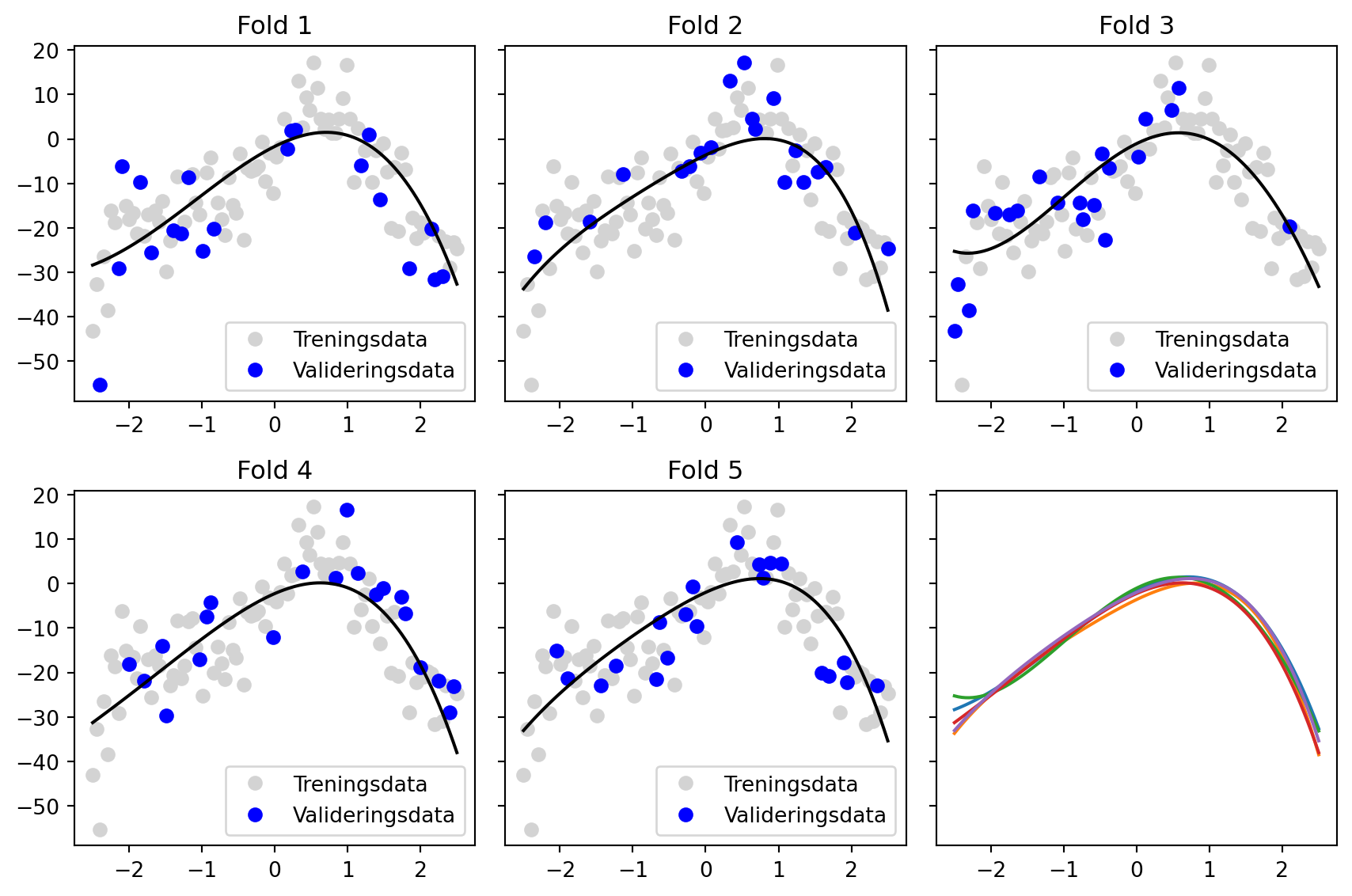
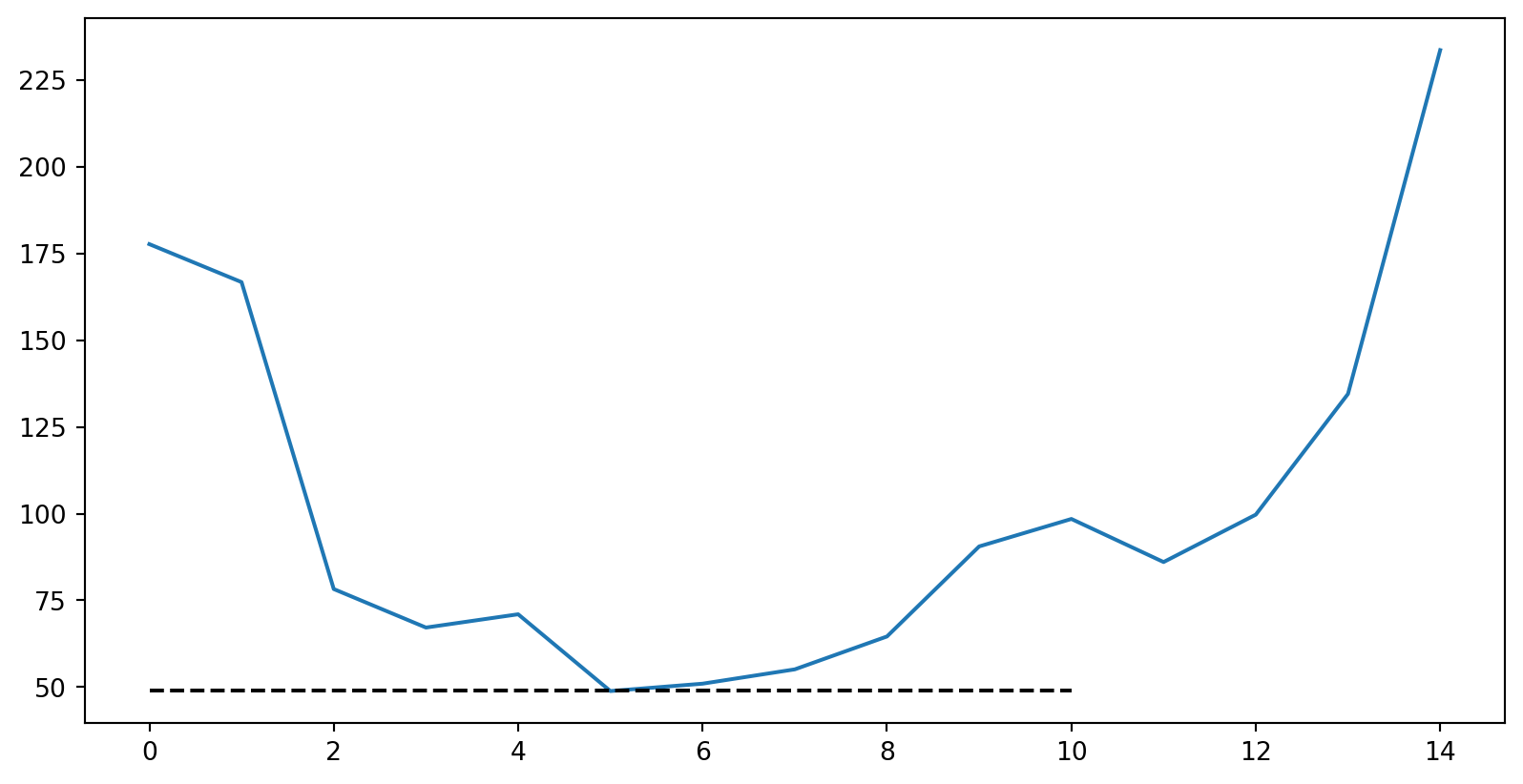
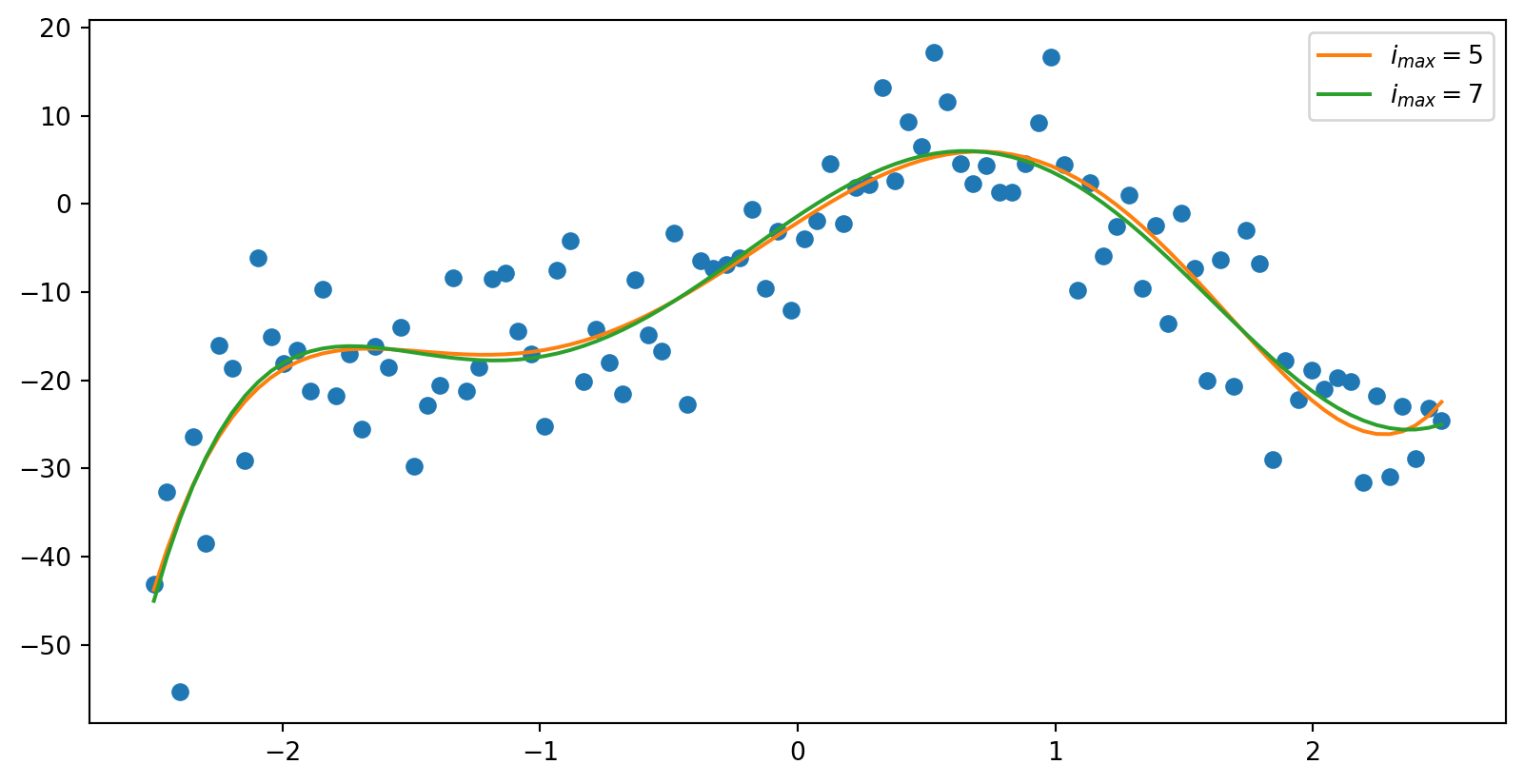
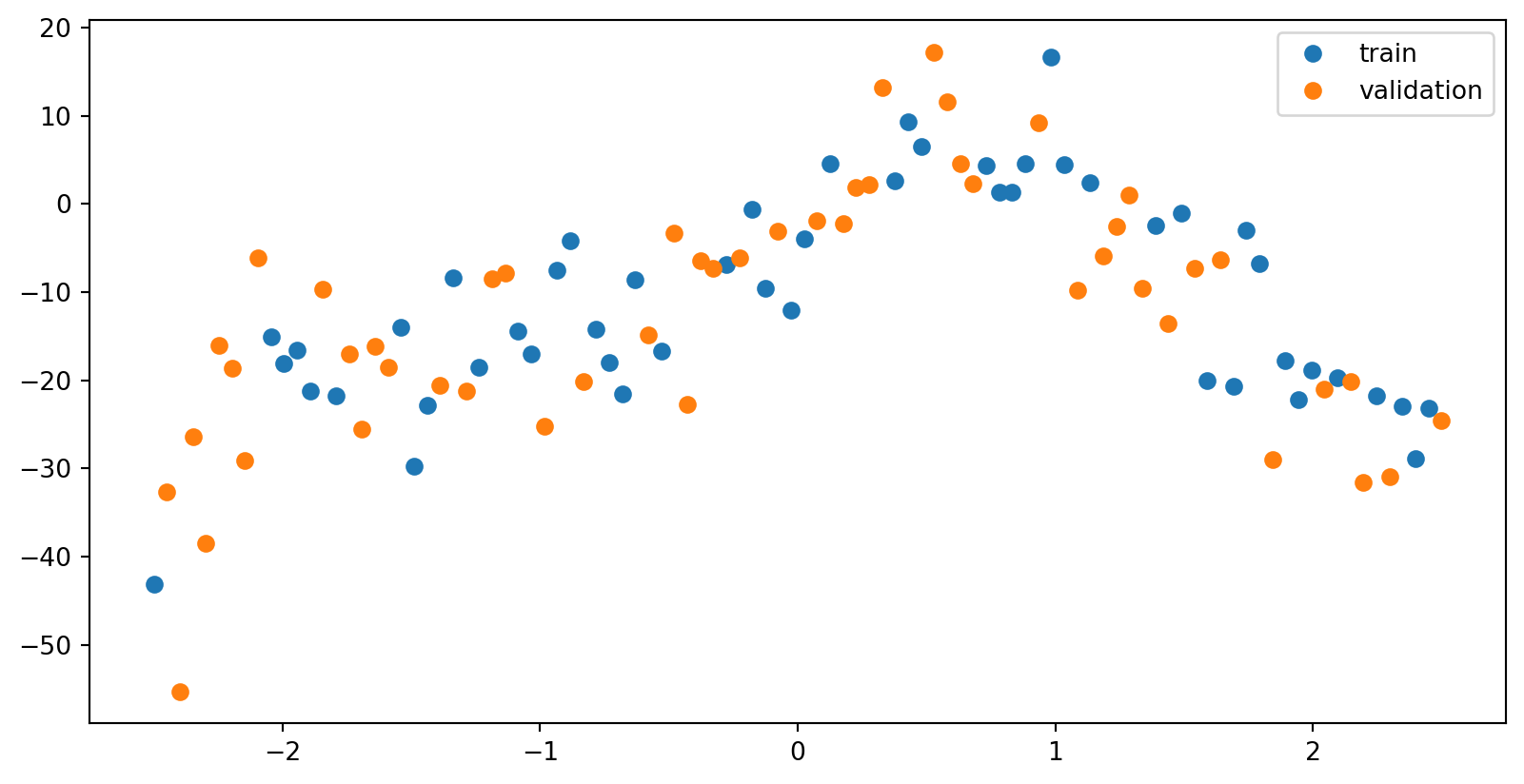
import numpy as np
data = np.loadtxt("data/hemmelig_funksjon.txt")
print(data)
x = data[:,0]
y = data[:,1][[-2.50000000e+00 -4.30998734e+01]
[-2.44949495e+00 -3.26987345e+01]
[-2.39898990e+00 -5.52854260e+01]
[-2.34848485e+00 -2.64284688e+01]
[-2.29797980e+00 -3.84960068e+01]
[-2.24747475e+00 -1.60790906e+01]
[-2.19696970e+00 -1.86770254e+01]
[-2.14646465e+00 -2.91149443e+01]
[-2.09595960e+00 -6.11836062e+00]
[-2.04545455e+00 -1.50227974e+01]
[-1.99494949e+00 -1.81119780e+01]
[-1.94444444e+00 -1.65905377e+01]
[-1.89393939e+00 -2.12568736e+01]
[-1.84343434e+00 -9.62635475e+00]
[-1.79292929e+00 -2.17751798e+01]
[-1.74242424e+00 -1.70026584e+01]
[-1.69191919e+00 -2.55251597e+01]
[-1.64141414e+00 -1.61060757e+01]
[-1.59090909e+00 -1.85088369e+01]
[-1.54040404e+00 -1.40123432e+01]
[-1.48989899e+00 -2.97722450e+01]
[-1.43939394e+00 -2.28487165e+01]
[-1.38888889e+00 -2.06196657e+01]
[-1.33838384e+00 -8.34302038e+00]
[-1.28787879e+00 -2.12597816e+01]
[-1.23737374e+00 -1.85375108e+01]
[-1.18686869e+00 -8.51863427e+00]
[-1.13636364e+00 -7.84281649e+00]
[-1.08585859e+00 -1.43893080e+01]
[-1.03535354e+00 -1.70080645e+01]
[-9.84848485e-01 -2.52287943e+01]
[-9.34343434e-01 -7.46827788e+00]
[-8.83838384e-01 -4.20727167e+00]
[-8.33333333e-01 -2.01172945e+01]
[-7.82828283e-01 -1.42435178e+01]
[-7.32323232e-01 -1.79813866e+01]
[-6.81818182e-01 -2.15525632e+01]
[-6.31313131e-01 -8.62162620e+00]
[-5.80808081e-01 -1.48933104e+01]
[-5.30303030e-01 -1.66653963e+01]
[-4.79797980e-01 -3.26441443e+00]
[-4.29292929e-01 -2.27294730e+01]
[-3.78787879e-01 -6.39051535e+00]
[-3.28282828e-01 -7.26030695e+00]
[-2.77777778e-01 -6.91538326e+00]
[-2.27272727e-01 -6.16544677e+00]
[-1.76767677e-01 -5.84873488e-01]
[-1.26262626e-01 -9.57800773e+00]
[-7.57575758e-02 -3.13795504e+00]
[-2.52525253e-02 -1.20919746e+01]
[ 2.52525253e-02 -3.97828128e+00]
[ 7.57575758e-02 -1.89130952e+00]
[ 1.26262626e-01 4.55607660e+00]
[ 1.76767677e-01 -2.20442195e+00]
[ 2.27272727e-01 1.85921687e+00]
[ 2.77777778e-01 2.15298119e+00]
[ 3.28282828e-01 1.31827583e+01]
[ 3.78787879e-01 2.67761036e+00]
[ 4.29292929e-01 9.30574969e+00]
[ 4.79797980e-01 6.47882568e+00]
[ 5.30303030e-01 1.72505807e+01]
[ 5.80808081e-01 1.15839155e+01]
[ 6.31313131e-01 4.53477786e+00]
[ 6.81818182e-01 2.25102486e+00]
[ 7.32323232e-01 4.33169718e+00]
[ 7.82828283e-01 1.31314261e+00]
[ 8.33333333e-01 1.35134770e+00]
[ 8.83838384e-01 4.61298306e+00]
[ 9.34343434e-01 9.23672856e+00]
[ 9.84848485e-01 1.66842812e+01]
[ 1.03535354e+00 4.48689914e+00]
[ 1.08585859e+00 -9.74590045e+00]
[ 1.13636364e+00 2.40751597e+00]
[ 1.18686869e+00 -5.86980559e+00]
[ 1.23737374e+00 -2.52396593e+00]
[ 1.28787879e+00 1.04290087e+00]
[ 1.33838384e+00 -9.60840164e+00]
[ 1.38888889e+00 -2.49422729e+00]
[ 1.43939394e+00 -1.35700524e+01]
[ 1.48989899e+00 -1.01926018e+00]
[ 1.54040404e+00 -7.29316159e+00]
[ 1.59090909e+00 -1.99989544e+01]
[ 1.64141414e+00 -6.33228306e+00]
[ 1.69191919e+00 -2.07056401e+01]
[ 1.74242424e+00 -3.01512622e+00]
[ 1.79292929e+00 -6.76592729e+00]
[ 1.84343434e+00 -2.90505187e+01]
[ 1.89393939e+00 -1.77320961e+01]
[ 1.94444444e+00 -2.22495263e+01]
[ 1.99494949e+00 -1.88432281e+01]
[ 2.04545455e+00 -2.10084218e+01]
[ 2.09595960e+00 -1.97315339e+01]
[ 2.14646465e+00 -2.01852203e+01]
[ 2.19696970e+00 -3.16092294e+01]
[ 2.24747475e+00 -2.18156289e+01]
[ 2.29797980e+00 -3.09277432e+01]
[ 2.34848485e+00 -2.29934136e+01]
[ 2.39898990e+00 -2.89183290e+01]
[ 2.44949495e+00 -2.31338756e+01]
[ 2.50000000e+00 -2.46218930e+01]]